今回はPydicomを使ってDICOMタグの取得をやってみたいと思います。
Contents
下準備
まだ、よくわからないという方のために詳しく説明していきたいと思います。
ある程度分かっている方は下準備は飛ばしてください
まずDICOM画像が手元にない場合は、Image-JのHPからサンプル画像がダウンロードできます。
拡張子(ファイル名の最後の文字)がdcmの画像がDICOM画像です。
ダウンロードした画像は、 デスクトップに「pydicom」というフォルダを作って入れておいてください。
(フォルダ名は何でも構いませんが、日本語のフォルダ名はプログラム実行時にエラーになってしまいますので必ず半角英数にて付けてください。)
続いて、コマンドプロンプトを立ち上げます。
そこに
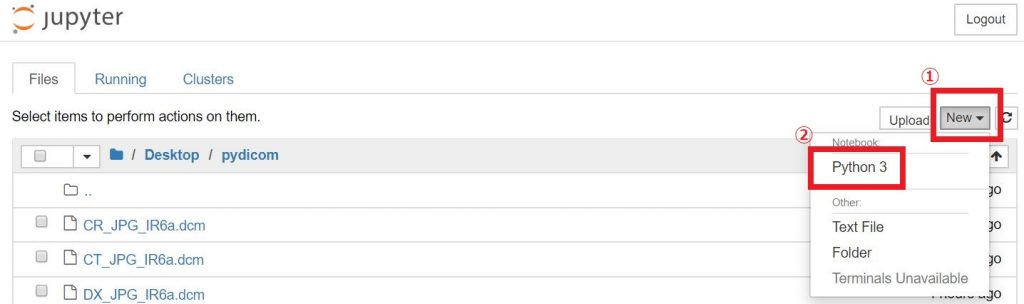
jupyter notebookと打ち込みます。少し無反応な時間が経ったのち、webブラウザが立ち上がり下図のページが立ち上がります。
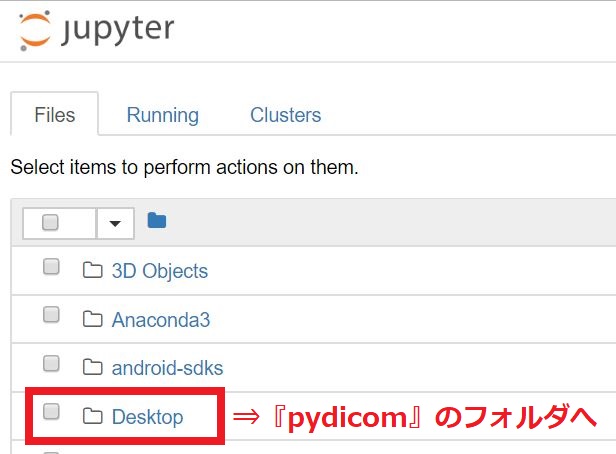
デスクトップをクリックし、pydicomのフォルダに移動します。
先ほど、ダウンロードした画像が入っていることが確認できます。
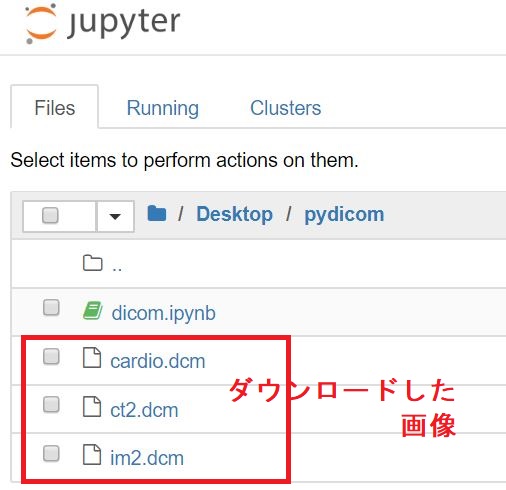
次に、このフォルダにコードを書いていくファイルを作成したいと思います。
画面右側上方に『New』というボタンがありますので
そちらをクリックし、『Python3』を選択します。
pythonコードのファイル名を決めます。
jupyterと書いてあるわきのUntitleをクリックします。
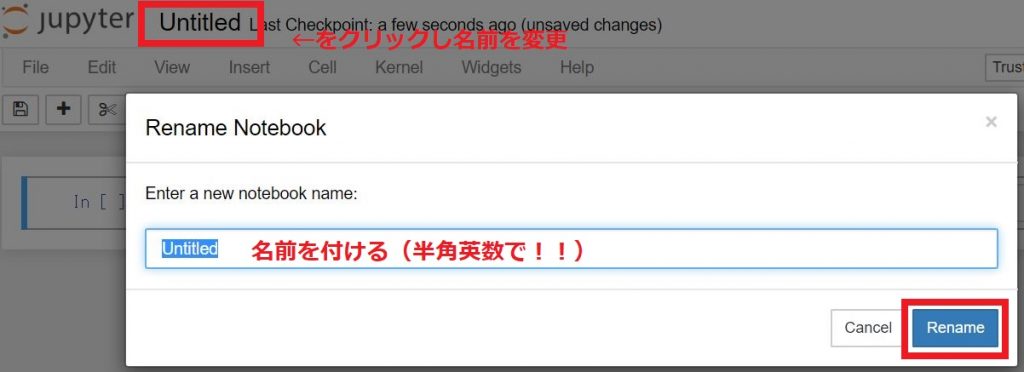
新しくい開いたダイアログに名前を付けて『Rename』をクリックします。
そうするとデスクトップにある『pydicom』のフォルダに
『先ほど付けた名前.ipynb』
のファイルが作成されていると思います。
ここまでできましたら、とりあえず下準備は完了です。
DICOMタグの表示
まず、簡単なところからDICOMタグを表示させてみましょう。
import pydicom
ds = pydicom.dcmread('CT2.dcm')
print(ds)コードが書けたら
shift + enterキーを押します。
いかがでしょうか?
下図のようにDICOMタグが表示できたと思います。
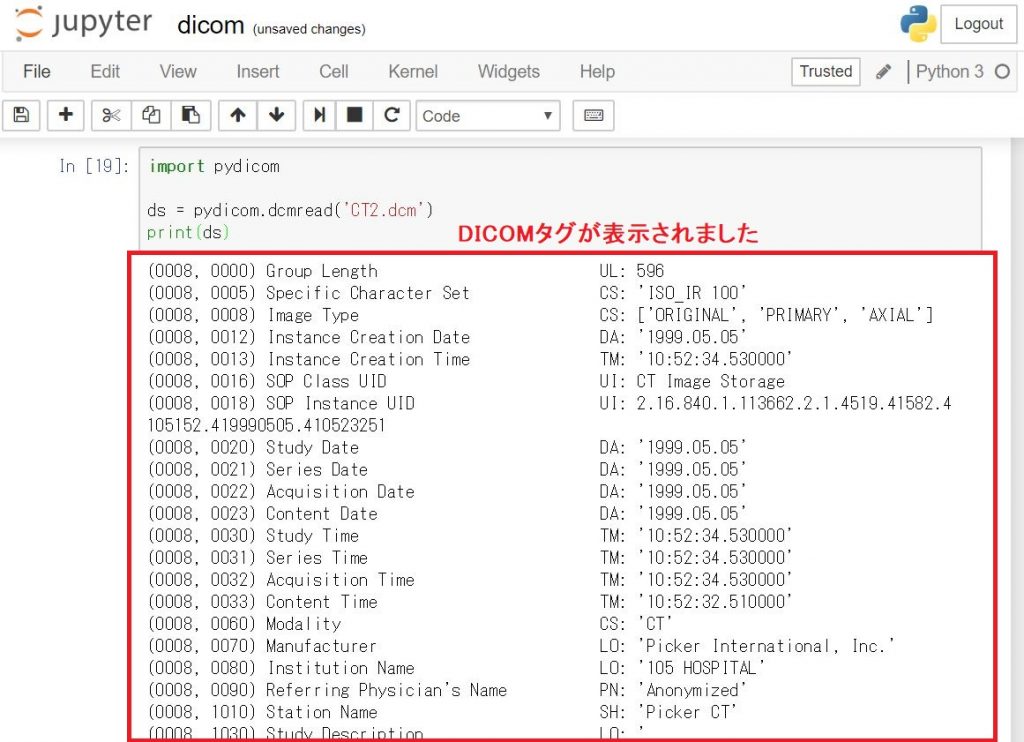
例えば、他のファイルのタグを見たい場合は上記コードの
ds = pydicom.dcmread(‘CT2.dcm‘)
下線部分に、ファイル名を指定すればOKです。
また、他のフォルダにあるファイルを指定したい場合はフルパスを指定することで表示することができます。
また、DICOMタグは項目ごとに表示することもできます。
例えば、モダリティーを見たい場合は
import pydicom
ds = pydicom.dcmread('CT2.dcm')
md = ds.Modality
print(md)で『CT』と表示させることもできます。
また、タグコードで指定することもできます。
コードは16進数に変換してあげる必要があるので、
16進数に変換する『0x』とコードの下2桁4桁を合わせて指定、それらコードを [ ] で囲み ます。
なので
モダリティーの場合は [0x0008,0x0060]
患者名の場合は [0x0010,0x0010]
を指定します。また、値だけど取得したい場合は
『.value』
を追加してあげます。指定しない場合はタグコードとタグ名、タグの値が返されます。
import pydicom
ds = pydicom.dcmread('CT2.dcm')
md = ds[0x08,0x60].value
print('ds[0x08,0x60].value ⇒ '+ str(md))
nm=ds[0x10,0x10].value
print('ds[0x10,0x10].value ⇒ '+ str(nm))
md = ds[0x08,0x60]
print('ds[0x08,0x60] ⇒ '+ str(md))
nm=ds[0x10,0x10]
print('ds[0x10,0x10] ⇒ '+ str(nm))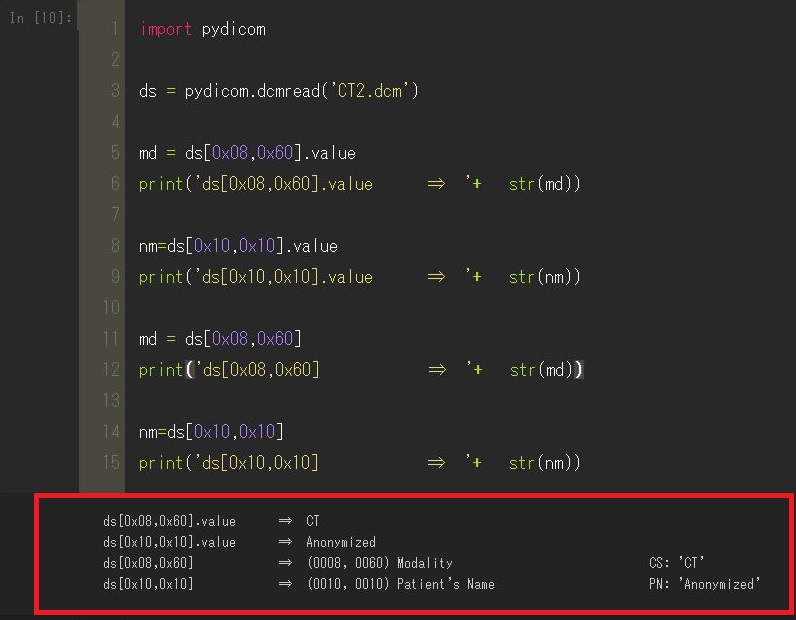
タグコードで指定する場合は、4桁で指定することができます!!
(画像が消えちゃっていたので追加したため背景色が変わっちゃっています・・・)
DICOMタグの取得は、 解像度を調べたり、撮影条件を調べたりといろいろと便利です。
検査の画像をシリーズで取得した場合、ファイル名がUIDでの表記になっていることがあり、その場合にファイル名を画像番号で名前変更したりと思った以上に使えますよ。