例えば、前立腺を抽出するセグメンテーションをしたい場合は、学習画像として、元画像と、前立腺だけを抽出した(下の画像右側)のような画像を作成しなければいけません。
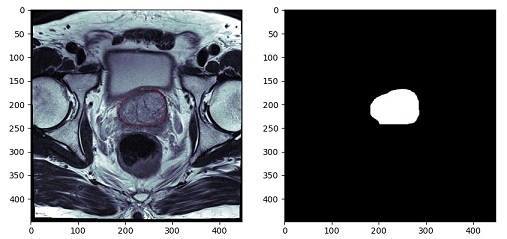
ワークステーションを使えばできるのですが、業務終了後に職場に残ってこの作業、やりたくないですよね。自分なら、家で酒でも飲みながら音楽をかけて作業。を望みます。
なので、今回は、この画像を作成するコードを組んでいきます。
コード
最終的なコードは以下となります。
import numpy as np
import cv2
import pydicom
import matplotlib.pyplot as plt
from matplotlib.patches import Circle
def on_motion(event):
global contour, fig, ax1
if event.button == 1 :
contour.append([int(event.xdata), int(event.ydata)])
ax1.add_patch(Circle((event.xdata, event.ydata), 0.1, color='r'))
fig.canvas.draw()
def on_button_release(event):
global contour, mask, ax2
cv2.fillConvexPoly(mask, np.array(contour), color=255, lineType=cv2.LINE_AA)
ax2.imshow(mask, cmap='bone')
fig.canvas.draw()
def main():
global contour, fig, ax1, ax2, mask
dcm = pydicom.dcmread('009_IMG11')
img = dcm.pixel_array
mask = np.zeros_like(img, dtype=np.uint8)
ww, wl = dcm[0x0028, 0x1051].value, dcm[0x0028, 0x1050].value
ww_l, ww_h = wl - ww // 2, wl + ww // 2
contour= []
fig = plt.figure(figsize=(10, 5))
ax1,ax2 = fig.add_subplot(1, 2, 1), fig.add_subplot(1, 2, 2)
ax1.imshow(img, cmap='bone', vmin=ww_l, vmax=ww_h)
fig.canvas.mpl_connect('button_release_event', on_button_release)
fig.canvas.mpl_connect('motion_notify_event', on_motion)
plt.show()
if __name__ == "__main__":
main()流れの説明
コードの流れとしては
- DICOM画像の表示
- マスク画像の作成(DICOM画像と同一の画像サイズで符号なしの8bit画像)
- DICOM画像で領域指定(右クリックを押したままマウス移動)
- マウスを移動させることでその座標をリストに追加していく
- マウスを離すことで領域抽出完了
- マスク画像に領域を描出
といった流れになっていきます。
上記コードで
DICOM画像の表示はmain関数内
DICOM画像で領域抽出は「on_motion」関数
マスク画像に領域を描出は「on_button_release」関数
で指定しています。
領域抽出
領域抽出は、DICOM画像上で抽出する領域をなぞっていくことでその座標を、リストに登録していきます。
ここでは、numpyの配列としてではなく、リストとして扱っていきます。
numpy配列として、2次元データに2次元データをひとつづつ追加していこうとするとちょっと厄介なことが起こるので。。。。。(詳細はいつか記事にしたいと思っています)
リストとして扱っていけば、純粋にappendで追加していけます。
追加する際に、整数型として追加していきます。
contour.append([int(event.xdata), int(event.ydata)])
また、なぞった位置をDICOM画像上に表示しておきたいので
ax1.add_patch(Circle((event.xdata, event.ydata), 0.1, color=’r’))
で画像上にプロットして
fig.canvas.draw()
で画像を再描出しています。
マスク画像に領域を描出 cv2.fillConvexPoly
なぞった座標のリストを別の画像上に描出します。
上記コードの32行目
mask = np.zeros_like(img, dtype=np.uint8)
でマスク画像用の配列をデータ0で作成しています。
配列の型は、マイナスデータなしの8bit画像です。なので階調は0~255となります。
先ほどなぞって登録したリストをmask上に描出します。
この機能は、openCVを使います。
cv2.fillConvexPoly(mask, np.array(contour), color=255, lineType=cv2.LINE_AA)
この関数は矩形を描出し、その中を塗りつぶします。
ここで先ほどの座標データを使うのですが、受けるデータはnumpy配列で整数型しか受けてくれません。
なので、リストに登録する際にint型を指定して追加していました。
そのリストをnumpy配列に変換するのは
np.array(contour)
としてあげるだけでnumpy配列として扱ってくれます。非常に便利です。
で、3番目の引数で255の値を指定してあげます。もし、RGBで指定するのであれば(0,0,0)等で指定してあげれば大丈夫です。ただ、その際にはmask配列作成時にRGBとして配列を作成することを忘れないようにしてください。
これで、先ほどなぞった座標内を255の値として登録することが出来ました。
後は、画像表示して確認です。
さいごに
いかがですか?このコードだけでは何枚もの教師画像を作成するのは大変ですが、根幹となるコードはできました。
このコードがどなたかの役に立てば幸いです。
The post 画像セグメンテーションに必要な教師データ作成ツールを組んでみた first appeared on 診療放射線技師がPythonをはじめました。.]]>やっと、深層学習の勉強に着手しました。
いきなりですが、U-NETでセグメンテーションをやってみようと思い、あるサイトからコードを完全コピペして動かしてみようとしたのですが「HDF5」のインポートでエラーが出て進まず困ったのでこの解決策を記事にしたいと思います。
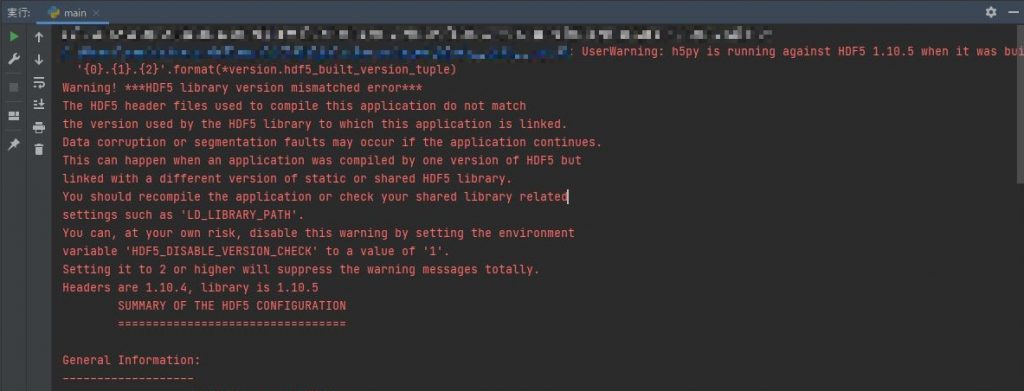
C:\Users\****\Anaconda3\envs\tf240\lib\site-packages\h5py__init__.py:40: UserWarning: h5py is running against HDF5 1.10.5 when it was built against 1.10.4, this may cause problems
‘{0}.{1}.{2}’.format(*version.hdf5_built_version_tuple)
Warning! HDF5 library version mismatched error
The HDF5 header files used to compile this application do not match
the version used by the HDF5 library to which this application is linked.
Data corruption or segmentation faults may occur if the application continues.
This can happen when an application was compiled by one version of HDF5 but
linked with a different version of static or shared HDF5 library.
You should recompile the application or check your shared library related
settings such as ‘LD_LIBRARY_PATH’.
You can, at your own risk, disable this warning by setting the environment
variable ‘HDF5_DISABLE_VERSION_CHECK’ to a value of ‘1’.
Setting it to 2 or higher will suppress the warning messages totally.
Headers are 1.10.4, library is 1.10.5
要約してみるとHDF5のヘッダーは1.10.4だけど、中身は1.10.5ですよ。
設定を変更することで強制的に実行できますよ
的な内容だと理解しました。(私の英語力では完全理解はできません・・・・)
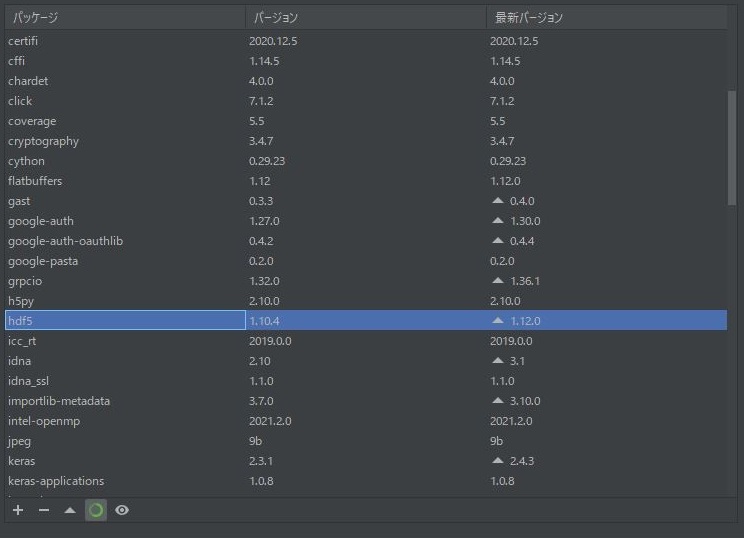
確かに、HDF5のバージョンは1.10.4になっています。
設定を変更しろと言ってもどこで変更すればいいのかわからない。
google先生に答えを求めると、一度削除後に再インストールで解決的なことが書いてあったので、ならばバージョンアップで対応できないかなと思ったのですが
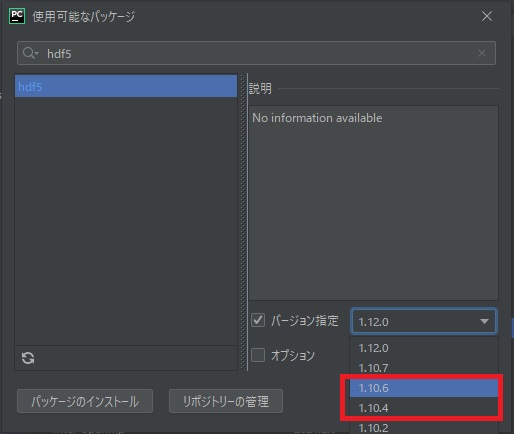
なぜ?1.10.5が無い・・・・
ならば、pipでインストール!!
まずは、アンインストールをして
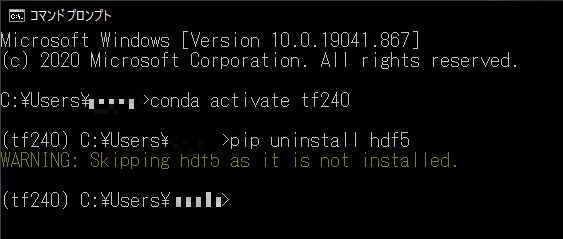
なぜか、インストールされていませんとのこと。。。。。
なので、Pycharmのインタープリタの画面からアンインストール
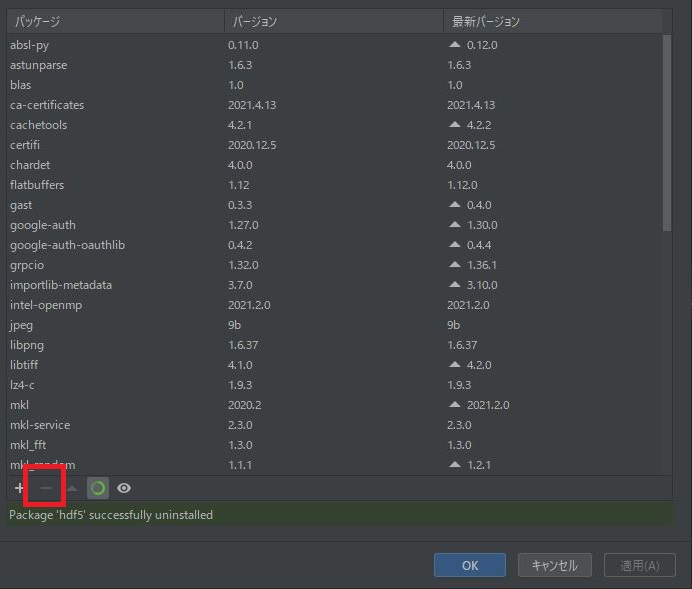
無事にアンインストールすることができました。
それでは、pipでインストール
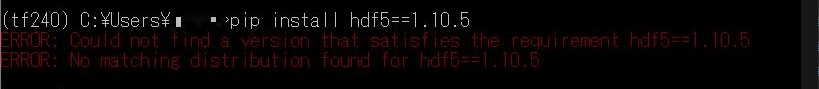
(tf240) C:\Users\****>pip install hdf5==1.10.5
ERROR: Could not find a version that satisfies the requirement hdf5==1.10.5
ERROR: No matching distribution found for hdf5==1.10.5
HDF5に1.10.5のバージョンは無いと怒られてしまいました。
どうしようか悩んでいたところ以下のサイトに解決策が載っていました。
pipでインストールできないのですが、condaコマンドでインストールができるみたいです。
また、conda-forgeというGit-Hubの組織があるらしく、そこには3000種類以上のパッケージがあるということです。
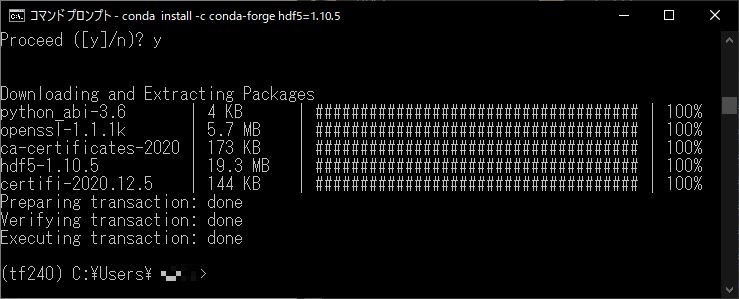
conda install -c conda-forge hdf5=1.10.5のコードを実行すると、以下の図のようにcondaのニューバージョンのお知らせと、HDF5とそれと依存関係のあるライブラリーもダウンロードしますよというお知らせが表示されますので、そのまま「Y」を入力しインストールを実行します。
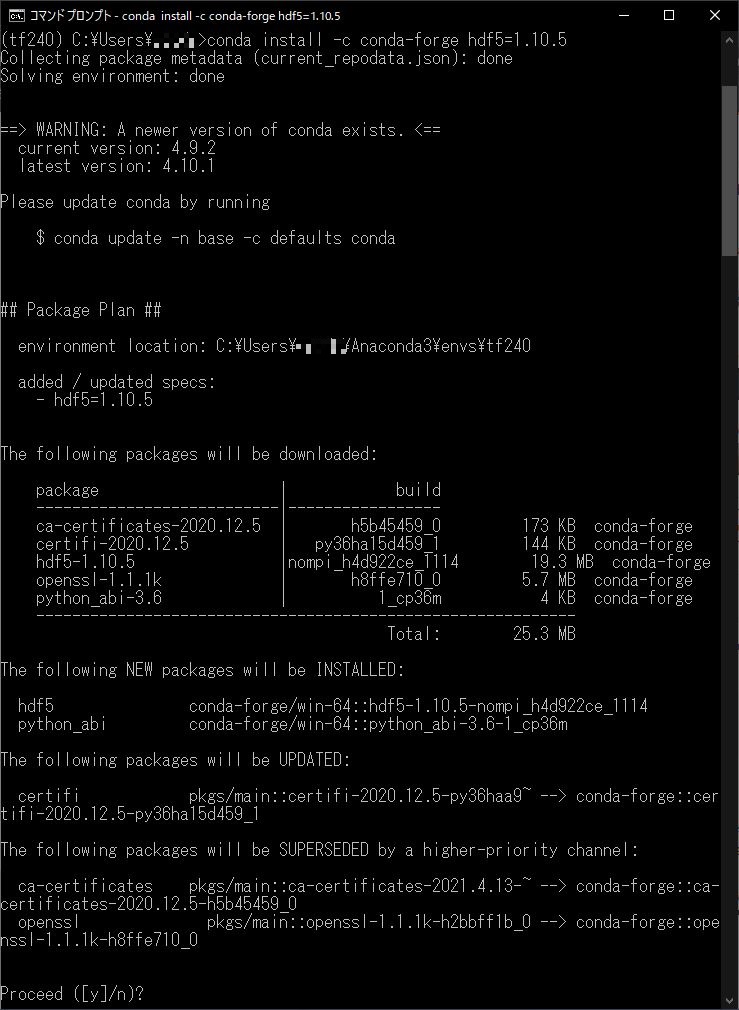
インストールが完了しました。
Pycharmで確認してみます。
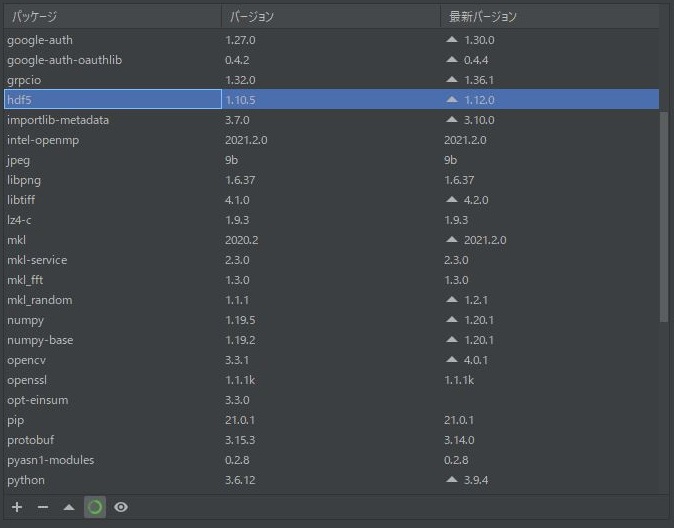
無事にHDF5のライブラリーをインストールすることができました。
これで、また一歩進みました。以上、HDF5バージョンミスマッチエラーの解決法でした。
お疲れさまでした。